Sự lưu hành và biến đổi di truyền của virus cúm A/H5N1 tại một số tỉnh vùng Đồng bằng sông Cửu Long
Nghiên cứu sự lưu hành và biến đổi di truyền của virus cúm A/H5N1 (viết tắt là CGC) đã được tiến
hành từ các mẫu dịch hầu họng (swab) lấy trên gà, vịt khỏe bán tại các chợ và các lò giết mổ tại một số
tỉnh Đồng bằng sông Cửu Long (An Giang, Kiên Giang, Đồng Tháp, thành phố Cần Thơ). Các mẫu được
xét nghiệm bằng kỹ thuật Realtime RT-PCR để định danh virus CGC và giải trình tự gene HA đối với một
số mẫu đại diện để xác định sự biến đổi di truyền và clade virus. Kết quả nghiên cứu cho thấy tỷ lệ dương
tính của virus CGC trên gia cầm tại 4 tỉnh ĐBSCL là 4,8%, trong đó tỷ lệ trên vịt là 5,5% và trên gà là
4,2%. Kết quả khảo sát sự biến đổi di truyền cho thấy các chủng virus CGC lưu hành tại địa phương có tỷ
lệ tương đồng ở mức độ nucleotide với tỷ lệ từ 98,2-99,3% và sai khác so với chủng virus khác phân lập
được ở Việt Nam và trên thế giới với tỷ lệ từ 3,7-10,3%. Trình tự các acid amin ở vị trí nối kết giữa đoạn
HA1 và HA2, đoạn quy định độc lực là RRRKR, tương đồng với trình tự của chủng virus CGC độc lực
cao tham chiếu A/chicken/Korea/IC546/2011(H5N1) và A/Hubei/1/2010(H5N1). Các chủng virus CGC
lưu hành tại các tỉnh An Giang và Kiên Giang thuộc clade virus 2.3.2.1c.

Trang 1

Trang 2
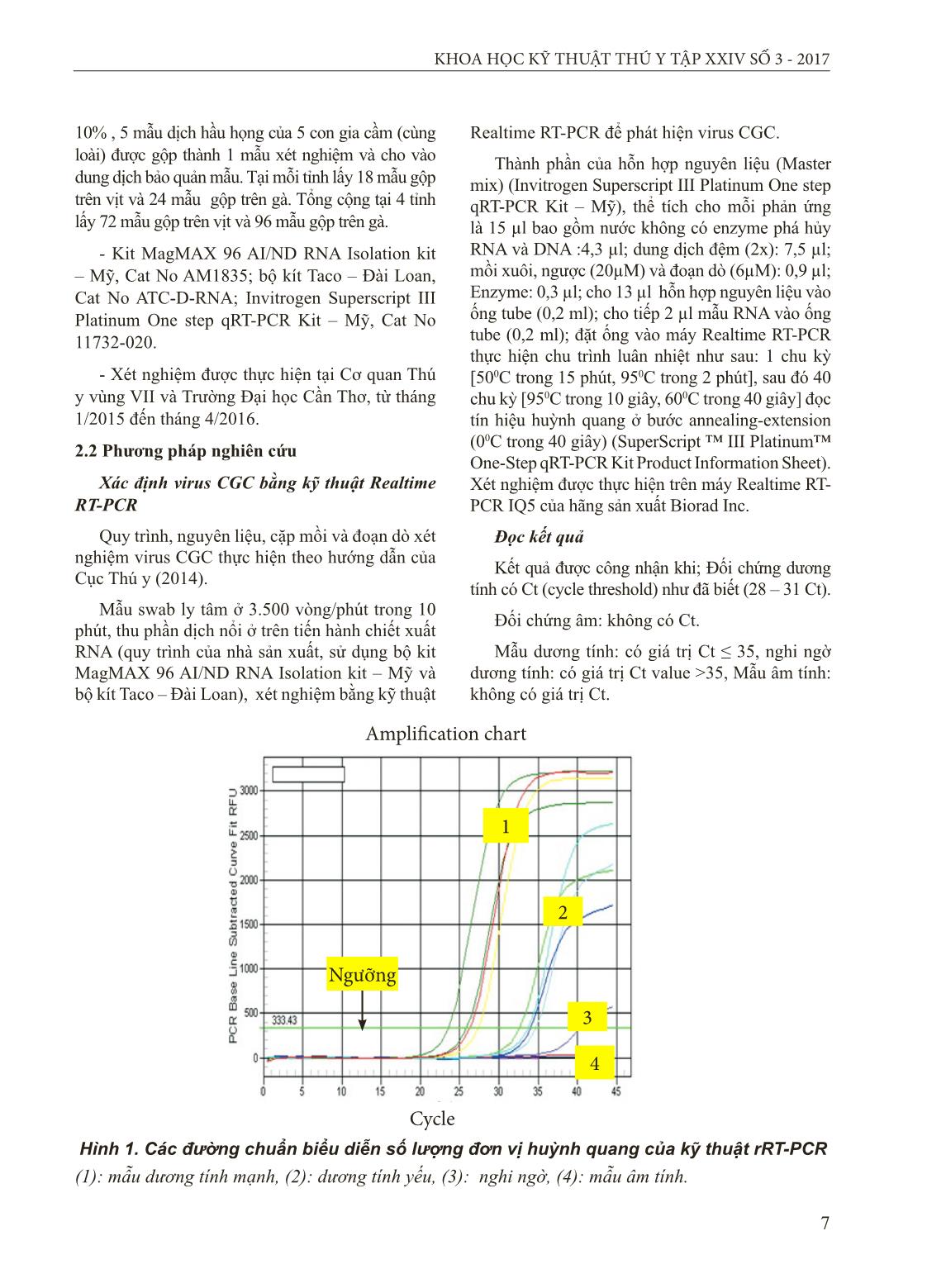
Trang 3

Trang 4
Trang 5
Trang 6
Trang 7

Trang 8

Trang 9
Tóm tắt nội dung tài liệu: Sự lưu hành và biến đổi di truyền của virus cúm A/H5N1 tại một số tỉnh vùng Đồng bằng sông Cửu Long

5KHOA HỌC KỸ THUẬT THÚ Y TẬP XXIV SỐ 3 - 2017 SÖÏ LÖU HAØNH VAØ BIEÁN ÑOÅI DI TRUYEÀN CUÛA VIRUS CUÙM A/H5N1 TAÏI MOÄT SOÁ TÆNH VUØNG ÑOÀNG BAÈNG SOÂNG CÖÛU LONG Tiền Ngọc Tiên, Phùng Thị Thanh Thúy, Nguyễn Quốc Hưng, Trần Diên Quý, Lý Thị Liên Khai Bộ môn Thú y, Khoa Nông nghiệp & SHUD - Trường Đại học Cần Thơ TÓM TẮT Nghiên cứu sự lưu hành và biến đổi di truyền của virus cúm A/H5N1 (viết tắt là CGC) đã được tiến hành từ các mẫu dịch hầu họng (swab) lấy trên gà, vịt khỏe bán tại các chợ và các lò giết mổ tại một số tỉnh Đồng bằng sông Cửu Long (An Giang, Kiên Giang, Đồng Tháp, thành phố Cần Thơ). Các mẫu được xét nghiệm bằng kỹ thuật Realtime RT-PCR để định danh virus CGC và giải trình tự gene HA đối với một số mẫu đại diện để xác định sự biến đổi di truyền và clade virus. Kết quả nghiên cứu cho thấy tỷ lệ dương tính của virus CGC trên gia cầm tại 4 tỉnh ĐBSCL là 4,8%, trong đó tỷ lệ trên vịt là 5,5% và trên gà là 4,2%. Kết quả khảo sát sự biến đổi di truyền cho thấy các chủng virus CGC lưu hành tại địa phương có tỷ lệ tương đồng ở mức độ nucleotide với tỷ lệ từ 98,2-99,3% và sai khác so với chủng virus khác phân lập được ở Việt Nam và trên thế giới với tỷ lệ từ 3,7-10,3%. Trình tự các acid amin ở vị trí nối kết giữa đoạn HA1 và HA2, đoạn quy định độc lực là RRRKR, tương đồng với trình tự của chủng virus CGC độc lực cao tham chiếu A/chicken/Korea/IC546/2011(H5N1) và A/Hubei/1/2010(H5N1). Các chủng virus CGC lưu hành tại các tỉnh An Giang và Kiên Giang thuộc clade virus 2.3.2.1c. Từ khóa: cúm gia cầm, virus A/H5N1, lưu hành, biến đổi di truyền, đồng bằng sông Cửu Long Circulation and genetic variation of A/H5N1 avian influenza virus on poultry in some Mekong delta provinces Tien Ngoc Tien, Phung Thi Thanh Thuy, Nguyen Quoc Hung, Tran Dien Quy, Ly Thi Lien Khai SUMMARY Avian influenza is an acute infectious disease caused by Orthomyxoviridae virus. Research on circulation and genetic variation of A/H5N1 avian influenza virus in 4 provinces of Mekong delta (An Giang, Kien Giang, Dong Thap provinces and Can Tho city) was conducted from the throat swab samples collecting from the healthy chickens and ducks at the live bird selling markets and slaughterhouses in the above mentioned provinces. The swab samples were tested by using Realtime RT-PCR technique to identify A/H5N1 avian influenza virus and to define the HA gene sequence so as to determine genetic variation and clade of virus. The studied result showed that the average prevalence of A/H5N1 avian influenza virus in poultry in the 4 Mekong delta provinces was 4.8%, of which the infection rate in duck was 5.5% and in chicken was 4.2%. Besides, result of investigation on genetic variation showed that nucleotide similarity rate between circulating virus strains in the 4 provinces was 98.2-99.3%, and the difference in comparison with other virus strains isolated in Viet Nam and the world was 3.7-10.3%. The amino acid sequence motif at connection position between HA1 and HA2 virulent segments was RRRK-R, and high similarity with the sequences of the studied virus strains and reference strain: A/chicken/Korea/IC546/2011(H5N1) and A/Hubei/1/2010(H5N1). The A/H5N1 avian influenza virus strains circulating in the provinces of Kien Giang and An Giang belonged to virus clade 2.3.2.1c. Keywords: avian influenza, virus A/H5N1, circulation, genetic variation, Mekong delta 6KHOA HỌC KỸ THUẬT THÚ Y TẬP XXIV SỐ 3 - 2017 1. ĐẶT VẤN ĐỀ Bệnh cúm gia cầm (Avian Influenza) là bệnh truyền nhiễm cấp tính của nhiều loài gia cầm, do virus cúm A thuộc họ Orthomyxoviridae gây ra.. Trong những tháng đầu năm 2016, các ổ dịch CGC đã xuất hiện tại 4 xã, phường của 4 huyện, thị xã thuộc 4 tỉnh/ thành phố (Bà Rịa-Vũng Tàu,Trà Vinh,TP Cần Thơ, Nghệ An). Số gia cầm mắc bệnh là 3046 con, số gia cầm tiêu hủy là 40.809 con (OIE, 2016). Theo WHO (2016), năm 2003 đã ghi nhận 3 trường hợp trên người bị nhiễm virus CGC và cả 3 ca bệnh đều tử vong. Trong những năm sau đó từ năm 2004 đến năm 2014, hằng năm tại Việt Nam đều có báo cáo ca bệnh CGC trên người. Đây là một bệnh truyền nhiễm cấp tính nguy hiểm gây tỷ lệ chết cao. Theo cảnh báo của tổ chức Y tế thế giới, nếu bệnh cúm gia cầm xảy ra trên diện rộng và kéo dài, virus cúm gia cầm có thể sẽ có sự biến đổi gene tạo ra chủng virus mới rất nguy hiểm đối với con người. Đồng bằng sông Cửu Long có ngành chăn nuôi gia cầm phát triển, đặc biệt là vịt được chăn nuôi phổ biến theo phương thức chạy đồng, nên nguy cơ xuất hiện các ổ dịch bệnh CGC là khá cao. Xuất phát từ thực tế trên, chúng tôi tiến hành nghiên cứu “Sự lưu hành và biến đổi di truyền của virus cúm gia cầm type A H5N1 trên gia cầm tại An Giang, Kiên Giang, Đồng Tháp và Thành phố Cần Thơ”. II. VẬT LIỆU VÀ PHƯƠNG PHÁP NGHIÊN CỨU 2.1 Vật liệu - Gà, vịt khỏe bán tại các chợ hoặc trong các lò giết mổ tại 4 địa phương trên. Mẫu dịch hầu họng (swab) được lấy ngẫu nh ... ct Information Sheet). Kiểm tra kích thước sản phẩm khuếch đại bằng phương pháp điện di trên thạch 2%. Sản phẩm khuếch đại của đoạn gene HA1 có kích thước 1100bp và HA2 có kích thước 1000bp. Chọn những mẫu có sản phẩm khuếch đại đoạn gene HA1 và HA2 đúng kích thước (1.100bp và 1000bp) theo thiết kế, gửi đi Công ty Macrogen, Hàn Quốc để giải trình tự gene. Phương pháp xử lý số liệu Các chuỗi trình tự đoạn gene HA của virus CGC được xử lý và phân tích bằng phần mềm Molecular Evolutionary Genetics Analysis (MEGA 6.0; Tamura et al, 2013). Sau khi có kết quả giải trình tự đoạn gene HA1 và HA2 sử dụng phần mềm Molecular Evolutionary Genetics Analysis (MEGA 6.0; Tamura et al, 2013) để liên kết, so sánh với các chủng tham chiếu từ ngân hàng gene bằng chức năng liên kết (Alignment by ClustalW), sau đó cắt bỏ những phần trùng lặp trình tự nucleotide giữa đoạn gene HA1 và HA2 và ghép nối hai đoạn lại với nhau thành một chuổi hoàn chỉnh; phân tích nhằm xác định sự biến đổi di truyền bằng chức năng liên kết (Alignment by ClustalW) và xác định phân nhóm virus cúm gia cầm type A H5N1 bằng phương pháp kết nối liền kề (Neighbor-Joining method) với hệ số Bootstrap 1.000 lần lặp lại. Các số liệu được xử lý bằng Fixer và Yates. III. KẾT QUẢ VÀ THẢO LUẬN 3.1 Kết quả khảo sát sự lưu hành của virus CGC trên gia cầm Kết quả được thể hiện ở bảng 2. Theo bảng 2 cho thấy tỷ lệ dương tính của virus CGC trên gia cầm tại các tỉnh An Giang và thành phố Cần Thơ là giống nhau (4,8%), trong khi đó tỉnh Kiên Giang có tỷ lệ cao hơn (9,5%), nhưng sự khác biệt này không có ý nghĩa thống kê (P (Ho) = 0,51224). Điều này có thể là do dịch bệnh CGC đã trở thành dịch địa phương (xảy ra hằng năm từ năm 2003 đến nay) tại các tỉnh vùng Đồng bằng sông Cửu Long nên virus CGC lưu hành rộng rãi trong quần thể gia cầm tại các địa phương. Đặc biệt tại tỉnh Đồng Tháp, kết quả nghiên cứu cho thấy không có sự lưu hành của virus CGC trên gia cầm trong thời gian khảo sát. Tỷ lệ dương tính tại các địa phương nằm trong khoảng từ 4,8 – 9,5% là thông tin cảnh báo khả năng gây ra các ổ dịch CGC, nhưng khả năng này không cao do tất cả các địa phương khảo sát trong nghiên cứu này đang triển khai chương trình tiêm phòng vacxin phòng bệnh cúm cho gia cầm. 9KHOA HỌC KỸ THUẬT THÚ Y TẬP XXIV SỐ 3 - 2017 Bảng 2. Tỷ lệ lưu hành của virus CGC trên gia cầm Tỉnh SMXN SMDT H5N1 Tỷ lệ (%) Vịt Gà SM XN SMDT H5N1 Tỷ lệ (%) SM XN SMDT H5N1 Tỷ lệ (%) An Giang 42 2 4,8 18 2 11,1 24 0 0 Kiên Giang 42 4 9,5 18 1 5,5 24 3 12,5 Đồng Tháp 42 0 0 18 0 0 24 0 0 TP. Cần Thơ 42 2 4,8 18 1 5,5 24 1 4,2 P (Ho) = 0,51224 EPT = 0,385714 EPT = 0,249645 Tổng 168 8 4,8 72 4 5,5 96 4 4,2 SMXN: Số mẫu xét nghiệm; SMDT: Số mẫu dương tính Tỷ lệ dương tính của virus CGC trong nghiên cứu này là 4,8-9,5% cao hơn so với kết quả của Dương Thị Thanh Thảo và Lý Thị Liên Khai (2010), theo đó, tỷ lệ dương tính tại Sóc Trăng là 1,67%. Bên cạnh đó, kết quả này cũng cao hơn rất nhiều so với kết quả nghiên cứu của Atanaska Marinova-Petkova & cs, 2014 tại Bangladesh là 0,33% Tỷ lệ dương tính của virus CGC trên vịt là 5,5%, trong đó, An Giang có tỷ lệ cao hơn (11,1%) so với các tỉnh còn lại, nhưng sự khác biệt này không có ý nghĩa thống kê (EPT = 0,385714). Tỷ lệ lưu hành trên gà tại Kiên Giang và thành phố Cần Thơ lần lượt là 12,5 % và 4,2%, nhưng sự khác biệt này không có ý nghĩa thống kê (EPT = 0,249645). Trong khi đó không phát hiện được sự lưu hành của virus CGC trên gà tại tỉnh An Giang và Đồng Tháp. Kết quả này tương đồng với kết quả nghiên cứu của Lưu Hữu Mãnh (2009) là không có lưu hành của virus CGC trên gà vào năm 2008 tại Hậu Giang và An Giang. 3.2 Kết quả giải trình tự gene HA và sự biến đổi di truyền của virus CGC lưu hành trên gia cầm Trong tổng số 8 mẫu có kết quả dương tính với virus CGC, tiến hành chọn một số mẫu đạt yêu cầu xét nghiệm để thực hiện giải trình tự gene HA. Kết quả đã giải trình tự được 4 mẫu, các đoạn gene HA được giải trình tự được trong nghiên cứu có chiều dài từ 1656 – 1695 nucleotide. Kết quả so sánh sự biến đổi thành phần nucleotide và acid amin của các chủng virus phân lập được trong nghiên cứu được trình bày trong bảng 3. Bảng 3. Số vị trí sai khác thành phần nucleotide (phía trên đường chéo) và acid amin (phía dưới đường chéo) so sánh giữa các chủng virus CGC phân lập được trong nghiên cứu Số lượng acid amin sai khác giữa các chủng virus CGC NC Số lượng nucleotide sai khác giữa các chủng virus CGC AA 1 2 3 4 1 21 26 22 2 4 29 25 3 8 10 12 4 4 6 6 1: A/Chick/KG/26/2015; 2: A/Chick/KG/34/2015; 3: A/Duck/AG/676/2015; 4: A/Duck/AG/677/2015 10 KHOA HỌC KỸ THUẬT THÚ Y TẬP XXIV SỐ 3 - 2017 Bảng 4. Sự biến đổi di truyền (nucleotide) của các chủng virus CGC trong nghiên cứu so sánh với chủng phân lập được ở Việt Nam và trên thế giới Ký hiệu Clade 1 2 3 4 Số vị trí biến sai khác Tỷ lệ (%) Số vị trí biến sai khác Tỷ lệ (%) Số vị trí biến sai khác Tỷ lệ (%) Số vị trí biến sai khác Tỷ lệ (%) A/Chicken/Vietnam/NCVD- 44/2007/H5N1 2.3.4 120 7,3 123 7,5 121 7,4 116 7,1 A/Barn swallow/Hong Kong/1161/2010/H5N1 2.3.2.1b 90 5,5 89 5,4 87 5,3 83 5,1 A/Chicken/Vietnam/NCVD- A937/2011/H5N1 1.1.2 160 9,8 159 9,7 160 9,8 156 9,5 A/Chicken/Vietnam/NCVD- swab-15/2008/H5N1 7.2 166 10,2 166 10,2 169 10,3 164 10 A/Duck/Cambodia/ PV027D1/2010/H5N1 1.1.2 150 9,2 153 9,3 150 9,2 146 8,9 A/Goose/Guangdong/1/1996 0 137 8,4 136 8,3 141 8,6 137 8,4 A/Hubei/1/2010 2.3.2.1a 61 3,7 62 3,9 61 3,7 57 3,5 A/chicken/Korea/IC546/2011 2.3.2.1c 61 3,7 65 4 65 4 61 3,7 A/Vietnam/1203/2004 1 121 7,4 124 7,6 124 7,6 120 7,3 1: A/Chick/KG/26/2015; 2: A/Chick/KG/34/2015; 3: A/Duck/AG/676/2015; 4: A/Duck/AG/677/2015 Kết quả bảng 3 cho thấy có sự khác biệt ở mức độ nucleotide và acid amin giữa các gene HA của virus H5N1 trong nghiên cứu. Số lượng các nucleotide sai khác nằm trong khoảng từ 12 - 29 (tỷ lệ tương đồng từ 98,2-99,3%). Chủng virus A/ Duck/AG/676/2015 (3) và A/Duck/AG/677/2015 (4) có sự sai lệch rất ít (12 nucleotide); cả hai chủng này đều được phát hiện tại tỉnh An Giang trong cùng một lần lấy mẫu. Trong khi đó, chủng virus A/Chick/KG/34/2015 lưu hành tại Kiên Giang có sự sai khác lớn hơn (29 nucleotide) so với các chủng virus lưu hành tại An Giang (A/ Duck/AG/676/2015). Đồng thời, kết quả so sánh sự khác biệt ở mức độ acid amin cho thấy có sự khác biệt giữa các chủng virus H5N1 từ 6 - 10 acid amin. Kết quả bảng 4 cho thấy các trình tự nucleotide của các chủng virus CGC lưu hành tại Kiên Giang và An Giang có sự khác biệt so với các chủng lưu hành ở Việt Nam và trên thế giới với tỷ lệ từ 3,7- 10,3%. Chủng virus lưu hành tại An Giang A/Duck/ AG/676/2015 có sự khác biệt so với chủng virus A/Chicken/Vietnam/NCVD-swab-15/2008/H5N1 với tỷ lệ cao nhất 10,3% (169 nucleotide), chủng virus A/Chicken/Vietnam/NCVD-swab-15/2008/ H5N1 thuộc clade 7.2 được phân lập trên gà vào năm 2008 tại miền Bắc Việt Nam và chủng virus này chỉ lưu hành trong khoảng thời gian rất ngắn ở miền Bắc, không phát tán ra các vùng, miền còn lại của Việt Nam (Nguyễn Tùng & cs, 2011). Bên cạnh đó kết quả so sánh cũng cho thấy các chủng virus CGC lưu hành tại An Giang và Kiên Giang có tỷ lệ tương đồng khá cao (96 – 96,3%) so với các chủng virus A/chicken/Korea/IC546/2011 (clade 2.3.2.1c) và A/Hubei/1/2010 (clade 2.3.2.1a). Điều này cho phép có thể dự đoán rằng các chủng virus trong nghiên cứu này sẽ thuộc clade 2.3.2.1c hoặc clade 2.3.2.1a. Ngoài ra, kết 11 KHOA HỌC KỸ THUẬT THÚ Y TẬP XXIV SỐ 3 - 2017 quả so sánh với chủng virus CGC gây bệnh trên người thuộc clade 1 phân lập được năm 2004 tại Việt Nam (A/Vietnam/1203/2004) đã cho thấy tỷ lệ khác biệt từ 7,3-7,6%, tương đương với sự khác biệt so với chủng virus phân lập trên gà vào năm 2007 thuộc clade 2.3.4 tại Việt Nam (A/Chicken/ Vietnam/NCVD-44/2007/H5N1). So sánh với các chủng virus CGC phân lập được tại Trung Quốc năm 1996 (A/Goose/Guangdong/1/1996) và Campuchia năm 2010 (A/Duck/Cambodia/ PV027D1/2010/H5N1) thì sự khác biệt chiếm tỷ lệ từ 8,3-9,3% (bảng 5). Kết quả phân tích trình tự các acid amin ở vị trí đoạn nối giữa HA1 và HA2 của các chủng virus CGC cho thấy các chủng A/Chick/KG/26/2015, A/Chick/KG/34/2015, A/Duck/AG/676/2015 và A/Duck/AG/677/2015 có trình tự giống với các chủng A/chicken/Vietnam/NCVD-44/2007-H5N1 Bảng 5. Trình tự acid amin đoạn nối giữa HA1 và HA2 (cleavage site) của các chủng virus CGC trong nghiên cứu so sánh với chủng phân lập được ở Việt Nam và trên thế giới Ký hiệu Trình tự acid amin đoạn nối giữa HA1 và HA2 (cleavage site) A/Chick/KG/26/2015 R R R K R A/Chick/KG/34/2015 R R R K R A/Duck/AG/676/2015 R R R K R A/Duck/AG/677/2015 R R R K R A/chicken/Vietnam/HU3-83/2015(H5N1) R R R K R A/duck/Vietnam/NCVD-15A60/2015(H5N1) R R R K R A/chicken/Khanhhoa/CVVI-27/2014(H5N1) R R R K R A/duck/Vietnam/NCVD14-A502/2014(H5N1) R R R K R A/chicken/Khanhhoa/CVVI-09/2013(H5N1) R R R K R A/muscovy_duck/Vietnam/NCVD-2074/2012(H5N1) R R R K R A/chicken/Korea/IC546/2011(H5N1) R R R K R A/Hubei/1/2010(H5N1) R R R K R A/Hong_Kong/6841/2010(H5N1) R R R K R A/chicken/Vietnam/NCVD-44/2007_H5N1 R R R K R A/chicken/Vietnam/NCVD-15A51/2015(H5N6) R R R K R A/chicken/Vietnam/NCVD14-A324/2014(H5N6) K R R K R A/muscovy_duck/Quang_Ninh/5c112/2013(H5N6) K R R K R A/chicken/Vietnam/NCVD-2716/2013(H5N1) R R K K R A/chicken/Soc_Trang/1/2012(H5N1) R R K K R A/chicken/Vietnam/NCVD-A937/2011(H5N1) R R K K R A/muscovy_duck/Vietnam/LBM66/2011_(H5N1) R R R K R A/barn_swallow/Hong_Kong/1161/2010(H5N1) R R R K R A/duck/Cambodia/PV027D1/2010(H5N1) R R K K R A/quail/Vietnam/1/2010(H5N1) R R K K R A/chicken/Vietnam/NCVD-swab15/2008_H5N1 R R R K R A/Viet_Nam/1203/2004 R R K K R A/goose/Guangdong/1/1996(H5N1) R R K K R 12 KHOA HỌC KỸ THUẬT THÚ Y TẬP XXIV SỐ 3 - 2017 (clade 2.3.4), A/Hubei/1/2010(H5N1) (clade 2.3.2.1a) và A/chicken/Korea/IC546/2011(H5N1) (clade 2.3.2.1c). Các chủng virus được phân lập vào khoảng thời gian dịch cúm gia cầm mới xảy ra như A/goose/Guangdong/1/1996 (H5N1) và A/ Viet_Nam/1203/2004 có trình tự acid amin giống nhau RRKKR, trong khi đó các chủng phân lập được vào khoảng thời gian từ năm 2007-2015 đã có sự thay đổi trình tự acid amin như chủng virus A/ barn_swallow/Hong_Kong/1161/2010(H5N1) và A/ chicken/Vietnam/NCVD-swab15/2008-H5N1 đã có sự thay đổi ở acid amin vị trí số 344 (Arginine (R) thay cho Lysine (K)). Các chủng virus cúm gia cầm type A H5N6 phân lập được trong những năm gần đây có sự khác biệt về trình tự ở vị trí nối giữa HA1 và HA2 so với virus cúm type A H5N1, acid amin ở vị trí 342 là Lysine (K) thay thế cho Arginine (R). 3.3 Kết qủa xác định clade virus CGC lưu hành trên gia cầm tại các địa phương nghiên cứu Để xác định clade virus CGC, sử dụng phần mềm MEGA 6.0 để phân tích và vẽ cây phả hệ. Bên cạnh các chủng virus đã giải trình tự trong nghiên cứu này, chúng tôi sử dụng thêm trình tự của các chủng virus tham chiếu (bảng 1) để vẽ cây phả hệ xác định clade virus. Kết quả được trình bày trong hình 2. Hình 2. Cây phả hệ các chủng virus CGC 2.3.2.1c 2.3.2.1a 2.3.2.1b 7.2 0 1 2.3.4 2.3.4.4 13 KHOA HỌC KỸ THUẬT THÚ Y TẬP XXIV SỐ 3 - 2017 Kết quả phân tích trình tự gene HA và vẽ cây phả hệ của các chủng virus CGC bằng phần mềm MEGA 6.0 (Tamura et al, 2013) đã xác định được tất cả các chủng virus đã giải trình tự đều thuộc clade virus 2.3.2.1c. Các chủng virus CGC lưu hành tại các tỉnh An Giang và Kiên Giang nằm cùng phân nhánh với chủng virus A/Hong Kong/6841/2010 (clade 2.3.2.1c) và A/chicken/Korea/IC546/2011 (clade 2.3.2.1c), điều này khẳng định các chủng virus này cũng thuộc clade 2.3.2.1c. Trong đó, các chủng virus lưu hành tại tỉnh An Giang là A/ Duck/AG/676/2015 và A/Duck/AG/677/2015 có mối quan hệ rất gần nhau (nằm cùng trong một phân nhánh), hai chủng virus còn lại lưu hành tại Kiên Giang A/Chick/KG/26/2015 và A/Chick/ KG/34/2015 thuộc hai phân nhánh khác nhau. IV. KẾT LUẬN - Tỷ lệ dương tính trung bình của virus CGC trên gia cầm tại một số tỉnh vùng đồng bằng sông Cửu Long (An Giang, Kiên Giang, Đồng Tháp, Thành phố Cần Thơ) là 4,8%. - Virus CGC lưu hành trên gà chiếm tỷ lệ 4,2% và trên vịt là 5,5%. - Các chủng virus CGC lưu hành tại các địa phương tương đồng ở mức độ nucleotide với tỷ lệ 98,2-99,3%. - Tỷ lệ nucleotide của các chủng virus CGC lưu hành tại các địa phương sai khác so với các chủng virus khác phân lập được ở Việt Nam và trên thế giới từ 3,7-10,3%. - Trình tự các acid amin ở vị trí nối kết giữa đoạn HA1 và HA2 (đoạn quy định độc lực) là RRRKR, tương đồng với trình tự của chủng virus CGC độc lực cao tham chiếu A/chicken/Korea/ IC546/2011(H5N1) và A/Hubei/1/2010(H5N1). - Các chủng virus CGC lưu hành tại các tỉnh An Giang và Kiên Giang thuộc clade virus 2.3.2.1c. TÀI LIỆU THAM KHẢO 1. Atanaska Marinova-Petkova, Mohammed M Feeroz, SM Rabiul Alam, M Kamrul Hasan, Sharmin Akhtar, Lisa Jones-Engel, David Walker, Laura McClenaghan, Adam Rubrum, John Franks, Patrick Seiler,Trushar Jeevan, Pamela McKenzie, Scott Krauss, Richard J Webby, and Robert G Webster, 2014. Multiple introductions of highly pathogenic avian influenza H5N1 viruses into Bangladesh. Emerging Microbes and Infections (2014) 3. 2. Dương Thị Thanh Thảo , Lý Thị Liên Khai, 2011. Khảo sát sự lưu hành và bước đầu giải trình tự gene của virus cúm gia cầm subtype H5N1 tại tỉnh Cà Mau và Sóc Trăng. Tạp chí Khoa học, Trường Đại học Cần Thơ, 2011:20a 7-17. 3. Lưu Hữu Mãnh, Nguyễn Bá Thành, Trương Thị Kim Dung, Đặng Thanh Tùng, Nguyễn Hiền Trung, Xầm Văn Lang, Chau Bora, Nguyễn Thị Thanh Tâm, 2009. Một số kết quả nghiên cứu về bệnh cúm gia cầm (Avian Influenza) ở Đồng bằng sông Cửu Long. Tạp chí khoa học Trường Đại học Cần Thơ, 11, 237-245. 4. Nguyễn Tùng, Đỗ Thị Hoa, Ngô Thị Thu Hương, Nguyễn Văn Cảm, Nguyễn Bá Hiên, 2011. Xác định một số đặc tính virus cúm gia cầm độc lực cao H5N1 nhánh 7 phân lập ở Việt Nam. Tạp chí Khoa học kỹ thuật Thú y số 2 năm 2011 5. Tamura K, Stecher G, Peterson D, Filipski A, and Kumar S (2013) MEGA6: Molecular Evolutionary Genetics Analysis version 6.0. Molecular Biology and Evolution:30 2725- 2729. 6. WHO/OIE/FAO, H5N1 Evolution Working Group (2014) Revised and updated nomenclature for highly pathogenic avian influenza A (H5N1) viruses. Influenza and Other Respiratory Viruses 8(3), 384–388. 7. www.oie.int/fileadmin/Home/eng/Health_ standards/tahm/2.03.04_AI.pdf 8. www.oie.int/en/animal-health-in-the-world/ update-on-avian-influenza/2016 9. www.who.int/influenza/human_animal_ interface/EN_GIP_201503031cumulative Number H5N1cases.pdf. Nhận ngày 28-11-2016 Phản biện ngày 26-2-2017
File đính kèm:
 su_luu_hanh_va_bien_doi_di_truyen_cua_virus_cum_ah5n1_tai_mo.pdf
su_luu_hanh_va_bien_doi_di_truyen_cua_virus_cum_ah5n1_tai_mo.pdf

